Virus dell'epatite E
| Orthohepevirus A | |
|---|---|
 | |
| Classificazione scientifica | |
| Dominio | Riboviria |
| Regno | Orthornavirae |
| Phylum | Kitrinoviricota |
| Classe | Alsuviricetes |
| Ordine | Hepelivirales |
| Famiglia | Hepeviridae |
| Genere | Orthohepevirus |
| Specie | Orthohepevirus A |
| Nomi comuni | |
|
Virus dell'epatite E | |
Il virus dell'epatite E (HEV) è l'agente eziologico dell'epatite E. Appartiene alla specie Orthohepevirus A. Un precedente nome della specie è stato Hepatitis E virus.[1][2]
Il carico globale di infezioni dai due principali genotipi (1 e 2) è stimato in 20 milioni all'anno, che provocano 70.000 morti e 3.000 aborti.[3]
La particella virale è stata osservata per la prima volta nel 1983[4] ma è stata clonata molecolarmente solo nel 1989.[5]
Tassonomia
[modifica | modifica wikitesto]Precedentemente era classificato nella famiglia Caliciviridae. Tuttavia, il suo genoma ricorda più da vicino il virus della rosolia. Ora è classificato come membro del genere Orthohepevirus nella famiglia Hepeviridae.[2]
Struttura
[modifica | modifica wikitesto]Le particelle virali hanno un diametro compreso tra 27 e 34 nanometri e non sono provviste di pericapside.[2][4]
Genoma e proteoma
[modifica | modifica wikitesto]Orthohepevirus A può essere classificato in otto diversi genotipi, facenti capo a diverse regioni geografiche: genotipo 1 (Asia), genotipo 2 (Africa e Messico), genotipo 3 (Europa e Nord America), genotipo 4 (Asia); i genotipi 5 e 6 sono stati rilevati nel cinghiale asiatico e i genotipi 7 e 8 nei cammelli.[2][6]
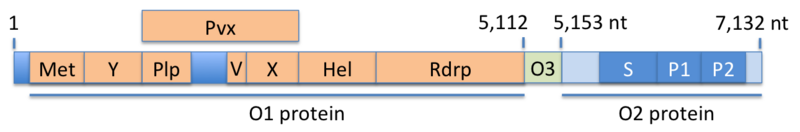
Il genoma virale consiste in un singolo filamento di RNA a senso positivo lungo circa 7200 basi. I tre frame a lettura aperta (ORF1, ORF2 e ORF3) codificano per tre proteine (O1, O2, O3), due delle quali sono poliproteine, cioè sono scisse in frammenti che svolgono le funzioni effettive del virus. La proteina O1 è costituita da sette di questi frammenti, vale a dire Met (metiltransferasi), Y (dominio Y), Plp (proteasi simile alla papaina), V (regione variabile ricca di prolina), X (dominio X, macro dominio), Hel (elicasi) e Rdrp (RNA polimerasi RNA-dipendente). Il dominio Pvx è una proteina di fusione costituita dai domini Plp, V e X. La proteina O3 è codificata da un singolo frame di lettura aperta (ORF3). La proteina O2 codifica per il capside, che è composto da tre domini, vale a dire il dominio shell (S) e due domini sporgenti (P1, P2).[7] I numeri nella figura indicano le posizioni nella sequenza dell'RNA.
Interattoma
[modifica | modifica wikitesto]L'interattoma proteina-proteina tra le proteine di Orthohepevirus A è stato mappato nel 2015 da Osterman et al., i quali hanno trovato 25 interazioni tra le 10 proteine studiate. Quasi tutte queste interazioni (24) sono state considerate di "alta qualità".[8]
Evoluzione
[modifica | modifica wikitesto]I ceppi di HEV che esistono oggi potrebbero essere derivati da un antenato comune risalente a un'epoca tra i 536 e i 1344 anni fa.[9] Un'altra analisi ha datato l'origine dell'epatite E a circa 6000 anni fa, suggerendo che questo fosse associato all'addomesticamento dei suini.[10] Ad un certo punto, due cladi potrebbero essersi allontanati, una forma antropotropica e una forma enzootica, che successivamente si sono evolute nei genotipi 1 e 2 e nei genotipi 3 e 4, rispettivamente.[11]
Mentre il genotipo 2 rimane meno comunemente rilevato rispetto ad altri genotipi, le analisi evolutive genetiche suggeriscono che i genotipi 1, 3 e 4 si sono diffusi considerevolmente negli ultimi 100 anni.[12]
Note
[modifica | modifica wikitesto]- ^ (EN) Michael A. Purdy, New Classification Scheme for Hepeviridae (PDF), su International Committee on Taxonomy of Viruses (ICTV), June 2014. URL consultato il 1º maggio 2019.«The species Hepatitis E virus will be renamed Orthohepevirus A, and the species Avian hepatitis E virus will be renamed Orthohepevirus B.»
- ^ a b c d ictv.global, http://www.ictv.global/report/hepeviridae.
- ^ Rein, D. B., Stevens, G. A., Theaker, J., Wittenborn, J. S. & Wiersma, S. T. (2012) The global burden of hepatitis E virus genotypes 1 and 2 in 2005. Hepatology 55, 988–97
- ^ a b Evidence for a virus in non-A, non-B hepatitis transmitted via the fecal-oral route, in Intervirology, vol. 20, n. 1, 1983, pp. 23-31, DOI:10.1159/000149370, PMID 6409836.
- ^ Isolation of a cDNA from the virus responsible for enterically transmitted non-A, non-B hepatitis, in Science, vol. 247, n. 4948, 1990, pp. 1335-9, Bibcode:1990Sci...247.1335R, DOI:10.1126/science.2107574, PMID 2107574.
- ^ Schlauder, G. G. & Mushahwar, I. K. (2001) Genetic heterogeneity of hepatitis E virus. J Med Virol 65, 282–92
- ^ Molecular virology of hepatitis E virus, in Virus Res., vol. 161, n. 1, 2011, pp. 47-58, DOI:10.1016/j.virusres.2011.02.011, PMID 21345356.
- ^ The Hepatitis E virus intraviral interactome, in Sci Rep, vol. 5, 2015, p. 13872, Bibcode:2015NatSR...513872O, DOI:10.1038/srep13872, PMID 26463011.
- ^ (EN) Yury E. Khudyakov e Michael A. Purdy, Evolutionary History and Population Dynamics of Hepatitis E Virus, in PLOS ONE, vol. 5, n. 12, 17 dicembre 2010, pp. e14376, Bibcode:2010PLoSO...514376P, DOI:10.1371/journal.pone.0014376, ISSN 1932-6203, PMID 21203540.
- ^ Sarra Baha, Nouredine Behloul e Zhenzhen Liu, Comprehensive analysis of genetic and evolutionary features of the hepatitis E virus, in BMC Genomics, vol. 20, n. 1, 29 ottobre 2019, p. 790, DOI:10.1186/s12864-019-6100-8, ISSN 1471-2164, PMID 31664890.
- ^ Mirazo S, Mir D, Bello G, Ramos N, Musto H, Arbiza J, New insights into the hepatitis E virus genotype 3 phylodynamics and evolutionary history, in Infect Genet Evol, vol. 43, 2016, pp. 267-73, DOI:10.1016/j.meegid.2016.06.003, PMID 27264728.
- ^ Izopet J, Abravanel F, Dalton H, Nassim RK (2014) Hepatitis E Virus Infection. Clin Micro Reviews 27 (1) 116–138
Collegamenti esterni
[modifica | modifica wikitesto]- ictv.global, http://www.ictv.global/report/hepeviridae.
- (EN) Hepatitis E virus, in Medical Subject Headings (MeSH), National Library of Medicine, 2009.
- Copia archiviata, su viprbrc.org. URL consultato il 28 agosto 2020 (archiviato dall'url originale il 9 dicembre 2012).