Proteinstruktur
Strukturen för ett protein, kallat proteinstrukturen, är mycket viktig både för dess egenskaper och dess funktion. Proteinstrukturer är mycket komplicerade. Ribosomen tillverkar från början bara en lång kedja av aminosyror, som binds ihop en efter en av peptidbindningar. Processen som kedjan sedan genomgår för att finna den form i vilken proteinet kan fylla sin funktion är också mycket komplicerad och kallas proteinveckning.[1]
Strukturnivåer
[redigera | redigera wikitext]
Aminosyrakedjan (även kallad polypeptidkedjan) som ett protein består av viks till en specifik tredimensionell form. Den slutliga formen bestäms av ordningen på aminosyrorna. Formen som ett protein naturligt viks till kallas för dess naturliga tillstånd. Formen har en avgörande betydelse för proteinets funktion.
Biokemister talar om fyra nivåer av struktur för ett protein:
- Primärstruktur, ordningen på aminosyrorna
- Sekundärstruktur, vikningar och spiraler som uppstår av vätebindningar mellan olika kedjedelar.
- Tertiärstruktur, den övergripande formen hos en proteinmolekyl och förhållandet mellan de olika sekundärstrukturerna.
- Kvartärstruktur, flera polypeptidkedjor.
Proteinets primära struktur
[redigera | redigera wikitext]Den primära strukturen för proteiner är ordningen på aminosyrorna som finns med. Att bestämma ett proteins primära struktur är alltså att namnge varje aminosyra i ordning från början till slut. Denna struktur anger proteinets exakta kemiska uppbyggnad.
Konventionen för att bestämma ordningen på aminosyror är att N-terminalen (änden med en fri amingrupp) är till vänster och C-terminalen (änden med en fri karboxylgrupp) till höger. Den primära strukturen hålls ihop av kovalenta peptidbindningar som skapas under proteinsyntesen.
Medan primärstrukturen till stor del bestämmer proteinets tredimensionella form, är det mycket svårt att enbart med hjälp av primärstrukturen räkna ut sekundär- och tertiärstrukturen, eftersom det är så många processer som bidrar till att skapa dessa. Man kan dock gissa sig till strukturen för en del av kedjan om man vet strukturen för en liknande peptidkedja (homologistudier). Man kan också förutsäga sekundärstrukturen för vissa sekvenser med hjälp av simuleringar.
Proteinets sekundära struktur
[redigera | redigera wikitext]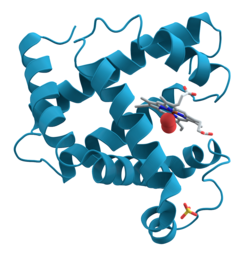
Växelverkningar mellan olika delar av aminosyrakedjan ger upphov till större, väldefinierade strukturer. Dessa strukturer utgör proteinets sekundärstruktur. Sekundärstrukturen visar hur olika delar av kedjan, en bit ifrån varandra, sitter ihop. Dock beskriver den inte deras faktiska tredimensionella positioner; detta behöver man tertiärstrukturen för att veta.
Det finns två huvudsakliga sekundärstrukturer: alfa-helixar och beta-flak, som båda uppstår på grund av vätebindningar. Egenskaperna hos primärstrukturen är en av de bidragande faktorerna som avgör vilka sekundärstrukturer som uppstår. En viss sekundärstruktur kan förekomma på många olika ställen i ett protein, och aminosyraavsnitt med helt olika primärstruktur kan ge upphov till liknande sekundärstrukturer.
Alfa-helixar
[redigera | redigera wikitext]Alfa-helixar är den vanligaste sekundärstrukturen. Aminosyrekedjan ”snurras” och skapar en spiral. Varje varv i denna spiral innehåller normalt 3,6 aminosyror. Skapandet av alfa-helixar är spontant och stabiliseras av vätebindningar. Olika aminosyror ingår i alfa-helixar med olika sannolikhet. I vissa fall är aminosyrornas sidokedjor (R-grupper) i vägen för varandra så att en alfa-helix inte kan bildas, så kallade steriska hinder. Aminosyran prolin ingår normalt inte i alfa-helixar på grund av dess begränsade rörlighet, medan aminosyran glycin å andra sidan är så extremt lättrörlig att den inte heller ofta deltar i alfa-helixar.
Beta-flak
[redigera | redigera wikitext]

Medan en alfa-helix består av en enskild linjär grupp av aminosyror så består betaflak av två eller fler olika delar av sträckor med åtminstone 5-10 aminosyror. Betaflak bildas av att olika delar av aminosyrekedjan binds till varandra. Vikningen och placeringen av sträckor av proteinets grundstomme (proteinet borträknat sidokedjorna) bredvid varandra för att forma beta-flaket skapas genom vätebindningar.
Sträckorna som beta-flaket består av kan antingen vara parallella eller anti-parallella. I parallella flak fortsätter närliggande peptidkedjor i samma riktning, medan i anti-parallella flak är närliggande kedjor arrangerade åt motsatt håll.
Loopar och random coils
[redigera | redigera wikitext]Alfa-helixar och beta-flak binds ihop av korta avsnitt av väldefinierade loopar, där aminosyrakedjan svänger tvärt, eller random coils, som är mer ostrukturerade. Det finns många olika loopstrukturer, som även de stabiliseras av vätebindningar.
Proteinets tertiära struktur
[redigera | redigera wikitext]Med ett proteins tertiära struktur menas den fullständiga tredimensionella strukturen av proteinet. Inkluderat i denna beskrivning är de rumsliga förhållandena olika sekundärstrukturer har till varandra inom en polypeptidkedja och hur dessa sekundärstrukturer själva hör ihop med den tredimensionella formen av proteinet.
Sekundärstrukturer av proteiner bildar ofta bestämda områden. Därför beskriver tertiärstrukturerna relationen av olika områden till varandra inom ett protein. Sådana sammansättningar av till exempel sekundärstruktur som är väldigt typiska och förekommer i många olika proteiner, kallas ibland för strukturmotiv (eng. Structural motif).
Denna struktur skapas av flera olika krafter. Den främsta kraften är hydrofoba interaktioner men vätebindningar, elektrostatiska interaktioner, van der Vaals-krafter och svavelbryggor spelar också en roll (se nedan).
Proteinets kvartära struktur
[redigera | redigera wikitext]
Många proteiner innehåller två eller flera proteinkedjor (polypeptidkedjor) som hålls ihop av samma icke-kovalenta krafter som stabiliserar tertiärstrukturerna i proteiner. De är alltså ihopsättningar av mer än en proteinmolekyl, eller monomer. Proteiner med flera proteinkedjor kallas för oligomeriska proteiner, eller multimerer.
Strukturen som formas av sammansättningen av de olika proteinkedjorna kallas för kvartärstrukturen. Oligomeriska proteiner kan bestå av flera identiska polypeptidkedjor eller flera olika sådana. Proteiner med identiska underenheter kallas homooligomerer, medan proteiner med flera olika polypeptidkedjor kallas för heterooligomerer.
Proteiner klassificeras vanligtvis i två större grupper:
- Fiberproteiner (eller fibrösa proteiner)
- Globulära proteiner
De fibrösa proteinerna kännetecknas av långa, raka kedjor. De är passiva strukturelement, i till exempel hår, naglar och bindevävnad). Fibrösa proteiner ger styrka och flexibilitet, är olösliga i vatten och har för det mesta bara en enda sekundärstruktur.
De globulära proteinerna bildar sfäriska molekyler och är "aktiva" proteiner (till exempel enzymer, transportproteiner). Globulära proteiner löser sig i vatten och har vanligtvis många olika sekundärstrukturer.
Krafter som kontrollerar proteinstrukturen
[redigera | redigera wikitext]Vätebindningar
[redigera | redigera wikitext]Polypeptider innehåller flera elektronegativa atomer både med och utan bundna väteatomer, både i grundstommarna och i R-grupperna (sidokedjorna). En elektronegativ atom som saknar ett väte kan bilda en vätebindning till en väteatom som är bunden till en annan elektronegativ atom, så att vätet i viss mån binds till båda de elektronegativa atomerna. En sådan elektronegativ atom är syre, som också finns tillsammans med väte i vattnet runt proteinet. Vätebindningar sker därför inte bara inom och mellan polypeptidkedjor utan också med det omgivande mediet. De andra två atomslagen som kan ge upphov till vätebindningar är fluor och kväve, varav den sistnämnda är ganska vanlig inom biokemin.
Hydrofoba krafter
[redigera | redigera wikitext]Proteiner består av aminosyror som innehåller antingen hydrofila eller hydrofoba R-grupper. En hydrofil (vattenälskande) grupp är polär och interagerar med fördel med vattnet och andra polära grupper, medan en hydrofob, vattenavstötande, grupp är opolär och energimässigt gynnas av att interagera med andra opolära grupper. Dessa interaktioner spelar den största rollen i formandet av proteinstrukturen. Hydrofoba grupper återfinns oftast på proteinets insida, där de inte interagerar med vattnet. Dessa krafter begränsar vilka möjliga former ett protein kan vikas till.
Elektrostatiska krafter
[redigera | redigera wikitext]Det finns även elektrostatiska krafter, det vill säga krafter mellan och inom laddningar och dipoler. Exempelvis understödjer reaktioner mellan olikt laddade R-grupper proteinveckning. Det sker även interaktioner mellan joniserade R-grupper och vattenmolekylen, som är en dipol, och även detta påverkar formen på proteinet.
van der Waals-krafter
[redigera | redigera wikitext]Det finns både attraherande och repulsiva Londonkrafter som bestämmer proteinvikning. Londonkrafter är mycket svaga, men det enorma antalet sådana interaktioner som finns i stora proteinmolekyler gör att de påverkar proteinets utformning.
Vad har strukturen för betydelse?
[redigera | redigera wikitext]Proteiner har olika funktioner att fylla, exempel på detta är att reagera med andra ämnen. Därför har formen stor betydelse för dess funktion. Om ett protein inte får den form som den skall ha för sin uppgift kommer proteinet inte att passa för det enzym den kodar för och därmed kommer reaktionen med andra ämnen antingen inte att ske överhuvudtaget eller inte på rätt sätt. Det är alltså formen på proteinet som ger den egenskaperna att kunna binda specifikt till vissa molekyler, exempelvis enzymer, och detta kan jämföras med en pusselbit där en korrekt formad protein kan passa in hos enzymet. De flesta enzymerna är dock flexibla och inte fasta vilket innebär att de kan anpassa sin form efter den reaktiva delen av proteinet som enzymet skall binda till. Beroende på hur aminosyrasekvensen ser ut får proteinet specifika funktioner den kan utföra. Aminosyrorna och miljön proteinet är syntetiserad i ger proteinet dess form.
Det finns många bioinformatiker och andra forskare som sysslar med att skapa proteiner på konstgjord väg. Dessa jobbar med att undersöka vilken form ett protein kommer att få eller vilket protein som behövs för att få en viss form.
Även om två proteiner har samma primärstruktur kan de få olika egenskaper om deras sekundär- och tertiärstrukturer är olika av någon anledning. Även om varje protein teoretiskt kan ha flera än en form, ses enbart en som dess ”riktiga” form, det vill säga den form som ger proteinets dess önskade egenskap.
Många problem kan uppstå av felaktiga proteinstrukturer. Utbytet av en hydrofobisk aminosyra (V) till en hydrofil aminosyra (E) i betakedjan av hemoglobin resulterar i sickle cell-anemi. Denna ändring av en enda aminosyra ändrar strukturen på enzymet så pass mycket att dess funktionalitet blir lidande, och personen som drabbas blir mycket sjuk. Ett annat exempel på felvikta proteiner är prioner som är skyldiga till bland annat galna ko-sjukan och Creutzfeldt–Jakobs sjukdom hos människor.
Med hjälp av kraftfulla datorer försöker man idag förutspå vilken form ett protein har i organismen utgående från dess primärstruktur. En forskare som vill skapa ett protein med vissa specifika egenskaper kan utgå från vilken form proteinet måste ha, och låta en dator räkna ut en primärstruktur som ger just den formen. För att öka beräkningshastigheten, och ge gemene man en möjlighet att hjälpa till vid forskningen har Stanford University startat projektet Folding@home, som bland annat forskar om Alzheimer, cancer och Parkinsons sjukdom
Se även
[redigera | redigera wikitext]Källor
[redigera | redigera wikitext]- King, Peptides and Proteins, Indiana State University School of Medicine
- Horton et al., Principles of Biochemistry, Pearson Education, Upper Saddle River 2006. ISBN 0-13-197736-9
- Folding at homes hemsida
Referenser
[redigera | redigera wikitext]- ^ ”The Shape and Structure of Proteins - Molecular Biology of the Cell - NCBI Bookshelf”. www.ncbi.nlm.nih.gov. https://www.ncbi.nlm.nih.gov/books/NBK26830/. Läst 3 april 2019.
