Poliovirus
 | |
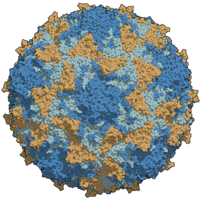 Kapsida poliovirusa tipa 3, obojeni bočni lanci proteina | |
| Sistematika | |
|---|---|
| Carstvo | Virusi |
| Koljeno | Enterovirusi |
| Razred | Enterovirusi |
| Red | RNK virusi |
| Porodica | Picornaviridae |
| Serotipovi | |
| |
Poliovirus, uzročnik poliomijelitisa (također poznat kao poliomijelitis), je serotip vrste Enterovirus C, u porodici Picornaviridae.[1] Postoje tri serotipa poliovirusa: tipovi 1, 2 i 3.
Poliovirus sastoji se od RNK genoma i proteinske kapside. Genom je jednolančana RNK pozitivnog smisla (+ssRNK) dug oko 7500 nukleotida.[2] Virusna čestica je prečnika oko 30 nm, sa ikosaedarnom simetrijom. Zbog svog kratkog genoma i jednostavnog sastava – samo RNK i bez omotača ikosaedarskog proteinskog omotača koji ga inkapsulira, poliovirus se naširoko smatra najjednostavnijim značajnim virusom.[3]
Poliovirus su prvi izolovali Karl Landsteiner i Erwin Popper, 1909.[4] Struktura virusa je prvi put razjašnjena 1958., pomoću difrakcije rendgenskih zraka u timu na Birkbeck College pod vođstvom Rosalind Franklin,[5][6] pokazujući da virus dječje paralize ima ikosaedarsku simetriju.[7]
Godine 1981. dva različita tima istraživača objavila su genom poliovirusa: Vincent Racaniello i David Baltimore na Massachusetts Institute of Technology (MIT)[8] i Naomi Kitamura i Eckard Wimmer na Stony Brook University.[9]
Trodimenzijsku strukturu poliovirusa odredio je 1985. godine James Hogle u Scripps Research Institute koristeći rendgensku kristalografiju.[10]
Poliovirus je jedan od najbolje okarakteriziranih virusa i postao je koristan model sistema za razumijevanje biologije RNK virusa.
Replikacijski ciklus
[uredi | uredi izvor]
Virion formira poru u ćelijskoj membrani kroz koju se virusna RNK oslobađa u citoplazmu (2).
Translacija virusne RNK se odvija putem IRES-posredovanog mehanizma (3). Poliprotein se cijepa, dajući zrele virusne proteine (4). RNK pozitivnog smisla služi kao šablon za komplementarnu sintezu negativnog lanca, proizvodeći dvolančani replikativni oblik (RF) RNK (5). Mnogo pozitivnih kopija RNK proizvodi se iz jednog negativnog lanca (6). Novosintetizovane molekule RNK pozitivnog smisla mogu poslužiti kao šabloni za translaciju više virusnih proteina (7) ili mogu biti zatvorene u kapsidu (8), koji na kraju stvara potomačke virione. Liza inficirane ćelije rezultira oslobađanjem viriona infektivnog potomstva (9).[11]
Poliovirus inficira ljudske ćelije vezujući se za receptor sličan imunoglobulinu, CD155 (također poznat kao receptor poliovirusa ili PVR)[12][13] on the cell surface.[14] Interakcija poliovirusa i CD155 olakšava ireverzibilnu konformacijsku promjenu virusne čestice neophodne za ulazak virusa.[15][16] Nakon vezivanja za ćelijsku membranu domaćina, smatralo se da se ulazak virusne nukleinske kiseline događa na jedan od dva načina: putem formiranja pore u plazmamebrani kroz koju se zatim RNK „ubrizgan” u citoplazmu ćeliju domaćina ili putem unosa virusa endocitozno posredovanim receptorom.[17] Recent experimental evidence supports the latter hypothesis and suggests that poliovirus binds to CD155 and is taken up by endocytosis. Immediately after internalization of the particle, the viral RNA is released.[18]
Poliovirus je pozitivnolančani RNK virus. Dakle, genom zatvoren unutar virusne čestice može se koristiti kao iRNK i odmah prevesti u ćeliji domaćina. Prilikom ulaska, virus otima ćelijski translacijski mehanizam, uzrokujući inhibiciju sinteze ćelijskih proteina u korist proizvodnje proteina specifične za virus.[19] Za razliku od iRNK ćelije domaćina, 5' kraj RNK poliovirusa je izuzetno duga—preko 700 nukleotida—i visoko strukturirana. Ovo područje virusnog genoma naziva se interno ulazno mjesto ribosoma (IRES). Ta regija sastoji se od mnogih sekundarnih struktura i tri ili četiri domena. Domen 3 je samosavijući RNK element koji sadrži konzervirane strukturne motive u različitim stabilnim petljama drške koje su povezane s dva četverosmjerna spoja. Kako se IRES sastoji od mnogih domena, ovi se sami sastoje od mnogih petlji koje doprinose modificiranom prevođenju bez 5' krajnjeg poklopca otimanjem ribosoma. Interakcijska petlja domena 3 poznata je kao GNRA tetrapetlja. Ostaci adenozina A180 i A181 u sekvenci GUAA tetrapetlje formiraju vodikove veze putem nekanonskih interakcija uparivanja baza sa parovima baza receptora C230/G242, odnosno G231/C241.[20] Genske mutacije u ovoj regiji sprečavaju proizvodnju virusnih proteina.[21][22][23] The first IRES to be discovered was found in poliovirus RNA.[24]
Informacijska RNK poliovirusa prevodi se kao jedan dugi polipeptid. Ovaj polipeptid se zatim autocijepa internim proteazama u oko 10 pojedinačnih virusnih proteina. Ne dešavaju se svi rascjepi sa istom efikasnošću. Stoga, količine proteina proizvedenih cijepanjem polipeptida variraju: naprimjer, proizvode se manje količine 3Dpol od onih kapsidnih proteina, VP1–4.[25][26] These individual viral proteins are:[3][27]

- 3Dpol, RNK ovisna RNK-polimeraza čija je funkcija da napravi više kopija virusnog RNK genoma
- 2Apro i 3Cpro/3CDpro, proteaze koje cijepaju virusni polipeptid
- VPg (3B), mali protein koji veže virusnu RNK i neophodan je za sintezu RNK pozitivnog i negativnog lanca virusa
- 2BC, 2B, 2C (na ATPazi)[28], 3AB, 3A, 3B proteini koji sadrže proteinski kompleks potreban za replikaciju virusa.
- VP0, koji se dalje cijepa na VP2 i VP4, VP1 i VP3, proteine virusnog kapsida
Nakon translacije, transkripcije i replikacije genoma koji uključuju jedan proces, ostvaruje se sinteza (+) RNK). Da bi se inficirajuća (+)RNK mogla replicirati, moraju biti transkribovane višestruke kopije (–)RNK i zatim korištene kao šabloni za sintezu (+)RNK. Replikativni intermedijari (RI), koji su asocijacija RNK molekula koji se sastoje od šablonske RNK i nekoliko rastućih RNK različite dužine, vide se u oba kompleksa replikacije za (–)RNK i (+)RNK. Za sintezu svakog negativnog i pozitivnog lanca RNK, kao prajmer VPg protein u poliovirusu djeluje RNK-ovisna RNK-polimeraza poliovirusa dodaje dva uracilska nukleotida (UU) VPg proteinu koristeći poli(A) rep na 3′-kraju +ssRNK genoma kao obrazac za sintezu antigenomske RNK negativnog lanca. Da bi se pokrenula ova sinteza – ssRNK, potreban je tirozin-hidroksil VPg. Ali za pokretanje sinteze pozitivnog lanca RNK potrebna je CRE-ovisna VPg uridililacija. To znači da se, kao prajmer, još jednom koristi VPg. ali ovaj put dodaje dva uridin-trifosfata koristeći cis-djelujući element replikacije (CRE) kao šablon.[29][30]
CRE poliovirusa identificiran je kao nepostignuta drška sa parom baza i konačna petlja koja se sastoji od 61 nukleotida. CRE se nalazi u enterovirusima. To je visoko konzerviran sekundarni strukturni element RNK i smješten u poliproteinskoj kodirajućoj regiji genoma. Kompleks se može translocirati u 5' regiju genoma koja nema aktivnost kodiranja, udaljenu najmanje 3,7 kb od početne lokacije. Ovaj proces može se odvijati bez negativnog uticaja na aktivnost. CRE kopije ne utiču negativno na replikaciju. Proces uridililacije VPg koji se odvija u CRE zahtijeva prisustvo 3CDpro, protein koji se vezuje za RNK. Povezuje se sa CRE direktno i posebno. Zbog svog prisustva VPg može pravilno vezati CRE i primarna proizvodnja se odvija bez problema.[31]
Neki od molekula (+) RNK koriste se kao šabloni za dalju sintezu (–)RNK, neki funkcionišu kao iRNK, a neki su predodređeni da budu genomi potomačkog viriona.[25]
U sklapanju novih virusnih čestica (tj. pakovanju potomačkih genoma u prokapsidu koji može preživjeti izvan ćelije domaćina), tj. uključujući:[32]
- Po pet kopija VP0, VP3 i VP1 čiji N-krajevi i VP4 čine unutrašnju površinu kapsida, sastavljaju se u 'pentamer' i 12 pentamera formira prokapsidu. (Spoljna površina kapsida sastoji se od VP1, VP2, VP3; C krajevi VP1 i VP3 koji formiraju usjeke oko svakog vrha; otprilike u to vreme, 60 kopija VP0 se cijepa u VP4 i VP2.)
- Svaka prokapsida dobija kopiju virusnog genoma, sa VPg i dalje pričvršćenim na 5' kraju.
Potpuno sastavljeni poliovirus lizom napušta granice svoje ćelije domaćina[33] 4 – 6 sati nakon iniciranja infekcije u kultiviranim ćelijama sisara.[34] Mehanizam oslobađanja virusa iz ćelije je nejasan,[2] ali svaka umiruća ćelija može osloboditi do 10.000 poliomijelitisnih viriona.[34]
Drake je pokazao da poliovirus može podvrgnuti višestrukoj reaktivaciji.[35] To jest, kada su poliovirusi ozračeni UV svjetlom i ostavljeni da se podvrgnu višestrukim infekcijama ćelija domaćina, održivo potomstvo moglo bi se formirati čak i pri dozama UV koje su inaktivirale virus u pojedinačnim infekcijama. Poliovirus može imati genetičke rekombinacije kada su najmanje dva virusna genoma prisutna u istoj ćeliji domaćinu. Kirkegaard i Baltimore[36] predstavili su dokaz da RNK-ovisna RNK- polimeraza (RdRP) katalizira rekombinaciju mehanizmom izbora kopije u kojem se RdRP prebacuje između (+)ssRNK šablona tokom sinteze negativnog lanca . Čini se da je rekombinacija u RNK virusima adaptivni mehanizam za popravku oštećenja genoma.[37][38]
Porijeklo i serotipovi
[uredi | uredi izvor]Poliovirus je strukturno sličan ostalim ljudskim enterovirusima (koksakivirusi, ehovirusi i rinovirusi), koji također koriste molekule slične imunoglobulinu za prepoznavanje i ulazak u ćelije domaćina.[13] Filogenetička analiza RNK i proteinskih sekvenci poliovirusa sugerira da je on možda evoluirao iz C-klastera Koksaki A virusa, zajedničkog pretka, koji je nastao mutacijom unutar kapsida.[39] Distinktna specijacija poliovirusa je vjerovatno nastala kao posljedica promjene specifičnosti ćelijskog receptora unutarćelijske adhezijske molekule-1 (ICAM-1), koju koriste C-klaster Koksaki A virusi, do CD155 ; što dovodi do promjene u patogenosti i omogućava virusu da inficira nervno tkivo.
Stopa mutacije u virusu je relativno visoka čak i za RNK viruse sa sinonimnom stopom supstitucije od 1,0 x 10−2 supstitucija/lokus/godina i neeksprimirana stopa zamjene od 3,0 x 10−4 zamjena/lokus/godina.[40] Bazna distribucija unutar genoma nije nasumična, pri čemu je adenozin rjeđi od očekivanog na 5' kraju i viši na 3' kraju.[41] Upotreba kodona nije nasumična, pri čemu se favoriziraju kodoni koji završavaju na adenozinu, a izbjegavaju se oni koji završavaju na citozinu ili guaninu. Upotreba kodona se razlikuje između tri genotipa i čini se da je vođena mutacijom, a ne selekcijom.[42]
Tri serotipa poliovirusa, PV-1, PV-2 i PV-3, svaki ima malo drugačiji kapsidni protein. Kapsidni proteini definiraju specifičnost ćelijskog receptora i antigenost virusa. PV-1 je najčešći oblik koji se sreće u prirodi, ali su sva tri oblika izuzetno zarazna.[4] Od marta 2020. divlji PV-1 je visoko lokaliziran u regijama u Pakistanu i Afganistanu. Certifikacija iskorjenjivanja autohtonog prijenosa dogodila se u septembru 2015. za divlji PV-2, nakon što je posljednji put otkriven 1999.,[43] iu oktobru 2019. za divlji PV-3, nakon što je posljednji put otkriven 2012. godine.[44]
Specifični sojevi svakog serotipa koriste se za pripremu vakcine protiv poliomijelitisa. Neaktivna poliomijelitisns vakcina priprema se formalinskom inaktivacijom tri divlja, virulentna referentna soja, Mahoney ili Brunenders (PV-1), MEF-1/Lansing (PV-2) i Saukett/Leon (PV-3) ). Oralna polio vakcina sadrži živa oslabljena (oslabljene) sojeve tri serotipa poliovirusa. Prolazni sojevi virusa u epitelnim ćelijama majnunskog bubrega uvode mutacije u virusni IRES i ometaju (ili slabe) sposobnost virusa da inficira nervno tkivo.[34]
Poliovirusi su ranije bili klasifikovani kao posebna vrsta koja pripada rodu Enterovirus u porodici Picornaviridae. U 2008., eliminis+rana je vrsta „poliovirus“ i tri serotipa su dodeljena vrsti „ljudski enterovirus C“ (kasnije preimenovanoj u „Enterovirus C“), u rodu „Enterovirus“ u porodici Picornaviridae. Tipska vrsta roda Enterovirus promijenjena je iz Poliovirus u (ljudski) Enterovirus C'.[45]
Kloniranje i sinteza
[uredi | uredi izvor]
Godine 1981. Racaniello i Baltimore koristili su tehnologiju rekombinantne DNK za stvaranje prvog infektivnog klona životinjskog RNK virusa, poliovirusa. DNK koja kodira RNA genom poliovirusa uvedena je u kultivirane ćelije sisara i proizveden je infektivni poliovirus.[46] Stvaranje infektivnog klona pokrenulo je razumijevanje biologije poliovirusa i postalo je standardna tehnologija koja se koristi za proučavanje mnogih drugih virusa.
Godine 2002, grupa Eckarda Wimmera na Stony Brook University uspjela je sintetizirati poliovirus iz njegovog hemijskog koda, proizvodeći prvi sintetski virus na svijetu.[47] Naučnici su prvo pretvorili objavljenu RNK sekvencu poliovirusa, dugu 7.741 bazu, u sekvencu DNK, jer je DNK bilo lakše sintetizirati. Kratki fragmenti ove sekvence dobijeni su poštom i sastavljeni. Kompletan virusni genom je zatim sastavila kompanija Gene synthesis. U sintetizovanu DNK ugrađeno je 19 markera, tako da se može razlikovati od prirodnog poliovirusa. Enzimi su korišteni za pretvaranje DNK nazad u RNK, njeno prirodno stanje. Drugi enzimi su zatim korišteni za prevođenje RNK u polipeptid, proizvodeći funkcionalnu virusnu česticu. Cijeli ovaj mukotrpan proces trajao je dvije godine. Novostvoreni sintetski virus ubrizgan je u PVR transgene miševe, kako bi se utvrdilo da li je sintetska verzija u stanju da izazove bolest. Sintetski virus je bio u stanju da se replicira, inficira i izazove paralizu ili smrt kod miševa. Međutim, sintetska verzija je bila između 1.000 i 10.000 puta slabija od originalnog virusa, vjerovatno zbog jednog od dodanih markera.[48]
Modifikacija za terapije
[uredi | uredi izvor]Modifikacija poliovirusa, zvana PVSRIPO, testirana je u ranim kliničkim ispitivanjima kao mogući lijek za rak.[49]
Reference
[uredi | uredi izvor]- ^ Ryan KJ, Ray CG, ured. (2004). Sherris Medical Microbiology (4th izd.). McGraw Hill. ISBN 978-0-8385-8529-0.
- ^ a b Hogle JM (2002). "Poliovirus cell entry: common structural themes in viral cell entry pathways". Annual Review of Microbiology. 56: 677–702. doi:10.1146/annurev.micro.56.012302.160757. PMC 1500891. PMID 12142481.
- ^ a b Goodsell DS (1998). The machinery of life. New York: Copernicus. ISBN 978-0-387-98273-1.Šablon:Page?
- ^ a b Paul JR (1971). A History of Poliomyelitis. (Yale studies in the history of science and medicine). New Haven, Conn: Yale University Press. ISBN 978-0-300-01324-5.Šablon:Page?
- ^ "Behind the picture: Rosalind Franklin and the polio model". Medical Research Council. 14. 3. 2019. Arhivirano s originala, 30. 10. 2018. Pristupljeno 4. 9. 2019.
- ^ Maddox B (2003). Rosalind Franklin: The Dark Lady of DNA. London: Harper Collins. str. 296. ISBN 0-00-655211-0.
- ^ Brown A (2007). J.D. Bernal: The Sage of Science. New York: Oxford University Press. str. 359–61. ISBN 978-0-19-920565-3.
- ^ Racaniello VR, Baltimore D (august 1981). "Molecular cloning of poliovirus cDNA and determination of the complete nucleotide sequence of the viral genome". Proceedings of the National Academy of Sciences of the United States of America. 78 (8): 4887–91. Bibcode:1981PNAS...78.4887R. doi:10.1073/pnas.78.8.4887. PMC 320284. PMID 6272282.
- ^ Kitamura N, Semler BL, Rothberg PG, Larsen GR, Adler CJ, Dorner AJ, et al. (juni 1981). "Primary structure, gene organization and polypeptide expression of poliovirus RNA". Nature. 291 (5816): 547–53. Bibcode:1981Natur.291..547K. doi:10.1038/291547a0. PMID 6264310. S2CID 4352308.
- ^ Hogle JM, Chow M, Filman DJ (27 Sep 1985). "Three-dimensional structure of poliovirus at 2.9 A resolution". Science. 229 (4720): 1358–65. doi:10.1126/science.2994218. PMID 2994218.
- ^ a b De Jesus NH (juli 2007). "Epidemics to eradication: the modern history of poliomyelitis". Virology Journal. 4 (1): 70. doi:10.1186/1743-422X-4-70. PMC 1947962. PMID 17623069.
- ^ Mendelsohn CL, Wimmer E, Racaniello VR (mart 1989). "Cellular receptor for poliovirus: molecular cloning, nucleotide sequence, and expression of a new member of the immunoglobulin superfamily". Cell. 56 (5): 855–65. doi:10.1016/0092-8674(89)90690-9. PMID 2538245. S2CID 44296539.
- ^ a b He Y, Mueller S, Chipman PR, Bator CM, Peng X, Bowman VD, et al. (april 2003). "Complexes of poliovirus serotypes with their common cellular receptor, CD155". Journal of Virology. 77 (8): 4827–35. doi:10.1128/JVI.77.8.4827-4835.2003. PMC 152153. PMID 12663789.
- ^ Dunnebacke TH, Levinthal JD, Williams RC (oktobar 1969). "Entry and release of poliovirus as observed by electron microscopy of cultured cells". Journal of Virology. 4 (4): 505–13. doi:10.1128/JVI.4.4.505-513.1969. PMC 375900. PMID 4309884.
- ^ Kaplan G, Freistadt MS, Racaniello VR (oktobar 1990). "Neutralization of poliovirus by cell receptors expressed in insect cells". Journal of Virology. 64 (10): 4697–702. doi:10.1128/JVI.64.10.4697-4702.1990. PMC 247955. PMID 2168959.
- ^ Gómez Yafal A, Kaplan G, Racaniello VR, Hogle JM (novembar 1993). "Characterization of poliovirus conformational alteration mediated by soluble cell receptors". Virology. 197 (1): 501–05. doi:10.1006/viro.1993.1621. PMID 8212594.
- ^ Mueller S, Wimmer E, Cello J (august 2005). "Poliovirus and poliomyelitis: a tale of guts, brains, and an accidental event". Virus Research. 111 (2): 175–93. doi:10.1016/j.virusres.2005.04.008. PMID 15885840.
- ^ Brandenburg B, Lee LY, Lakadamyali M, Rust MJ, Zhuang X, Hogle JM (juli 2007). "Imaging poliovirus entry in live cells". PLOS Biology. 5 (7): e183. doi:10.1371/journal.pbio.0050183. PMC 1914398. PMID 17622193.
- ^ Attardi G, Smith J (1962). "Virus specific protein and a ribo-nucleic acid associated with ribosomes in poliovirus infected HeLa cells". Cold Spring Harbor Symposia on Quantitative Biology. 27: 271–92. doi:10.1101/SQB.1962.027.001.026. PMID 13965389.
- ^ Lozano G, Fernandez N, Martinez-Salas E (februar 2016). "Modeling Three-Dimensional Structural Motifs of Viral IRES". Journal of Molecular Biology. 428 (5 Pt A): 767–76. doi:10.1016/j.jmb.2016.01.005. PMID 26778619.
- ^ Chen CY, Sarnow P (april 1995). "Initiation of protein synthesis by the eukaryotic translational apparatus on circular RNAs". Science. 268 (5209): 415–17. Bibcode:1995Sci...268..415C. doi:10.1126/science.7536344. PMID 7536344.
- ^ Pelletier J, Sonenberg N (juli 1988). "Internal initiation of translation of eukaryotic mRNA directed by a sequence derived from poliovirus RNA". Nature. 334 (6180): 320–25. Bibcode:1988Natur.334..320P. doi:10.1038/334320a0. PMID 2839775. S2CID 4327857.
- ^ Jang SK, Kräusslich HG, Nicklin MJ, Duke GM, Palmenberg AC, Wimmer E (august 1988). "A segment of the 5' nontranslated region of encephalomyocarditis virus RNA directs internal entry of ribosomes during in vitro translation". Journal of Virology. 62 (8): 2636–43. doi:10.1128/JVI.62.8.2636-2643.1988. PMC 253694. PMID 2839690.
- ^ Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. str. 4. ISBN 978-0-470-02386-0.
- ^ a b Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. str. 165. ISBN 978-0-470-02386-0.
- ^ Harper DR (2012). Viruses: Biology, Applications, and Control. The United States of America: Garland Science. ISBN 978-0-8153-4150-5.Šablon:Page?
- ^ "Poliomyelitis". Brown University. Arhivirano s originala, 22. 2. 2007.
- ^ Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. str. 164. ISBN 978-0-470-02386-0.
- ^ Louten J (2016). "Poliovirus". Essential Human Virology. Elsevier. str. 257–71. doi:10.1016/b978-0-12-800947-5.00014-4. ISBN 978-0-12-800947-5.
- ^ Murray KE, Barton DJ (april 2003). "Poliovirus CRE-dependent VPg uridylylation is required for positive-strand RNA synthesis but not for negative-strand RNA synthesis". Journal of Virology. 77 (8): 4739–50. doi:10.1128/JVI.77.8.4739-4750.2003. PMC 152113. PMID 12663781.
- ^ Goodfellow IG, Kerrigan D, Evans DJ (januar 2003). "Structure and function analysis of the poliovirus cis-acting replication element (CRE)". RNA. 9 (1): 124–37. doi:10.1261/rna.2950603. PMC 1370376. PMID 12554882.
- ^ Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. str. 161, 165. ISBN 978-0-470-02386-0.
- ^ Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. str. 166. ISBN 978-0-470-02386-0.
- ^ a b c Kew OM, Sutter RW, de Gourville EM, Dowdle WR, Pallansch MA (2005). "Vaccine-derived polioviruses and the endgame strategy for global polio eradication". Annual Review of Microbiology. 59: 587–635. doi:10.1146/annurev.micro.58.030603.123625. PMID 16153180.
- ^ Drake JW (august 1958). "Interference and multiplicity reactivation in polioviruses". Virology. 6 (1): 244–64. doi:10.1016/0042-6822(58)90073-4. PMID 13581529.
- ^ Kirkegaard K, Baltimore D (novembar 1986). "The mechanism of RNA recombination in poliovirus". Cell. 47 (3): 433–43. doi:10.1016/0092-8674(86)90600-8. PMC 7133339. PMID 3021340.
- ^ Barr JN, Fearns R (juni 2010). "How RNA viruses maintain their genome integrity". The Journal of General Virology. 91 (Pt 6): 1373–87. doi:10.1099/vir.0.020818-0. PMID 20335491.
- ^ Bernstein H, Bernstein C, Michod RE (januar 2018). "Sex in microbial pathogens". Infection, Genetics and Evolution. 57: 8–25. doi:10.1016/j.meegid.2017.10.024. PMID 29111273.
- ^ Jiang P, Faase JA, Toyoda H, Paul A, Wimmer E, Gorbalenya AE (maj 2007). "Evidence for emergence of diverse polioviruses from C-cluster coxsackie A viruses and implications for global poliovirus eradication". Proceedings of the National Academy of Sciences of the United States of America. 104 (22): 9457–62. Bibcode:2007PNAS..104.9457J. doi:10.1073/pnas.0700451104. PMC 1874223. PMID 17517601.
- ^ Jorba J, Campagnoli R, De L, Kew O (maj 2008). "Calibration of multiple poliovirus molecular clocks covering an extended evolutionary range". Journal of Virology. 82 (9): 4429–40. doi:10.1128/JVI.02354-07. PMC 2293050. PMID 18287242.
- ^ Rothberg PG, Wimmer E (decembar 1981). "Mononucleotide and dinucleotide frequencies, and codon usage in poliovirion RNA". Nucleic Acids Research. 9 (23): 6221–29. doi:10.1093/nar/9.23.6221. PMC 327599. PMID 6275352.
- ^ Zhang J, Wang M, Liu WQ, Zhou JH, Chen HT, Ma LN, et al. (mart 2011). "Analysis of codon usage and nucleotide composition bias in polioviruses". Virology Journal. 8: 146. doi:10.1186/1743-422X-8-146. PMC 3079669. PMID 21450075.
- ^ "Global eradication of wild poliovirus type 2 declared". Global Polio Eradication Initiative. 20. 9. 2015. Arhivirano s originala, 28. 9. 2015. Pristupljeno 30. 9. 2015.
- ^ "GPEI-Two out of three wild poliovirus strains eradicated" (jezik: engleski). Pristupljeno 2. 11. 2019.
- ^ Carstens EB, Ball LA (juli 2009). "Ratification vote on taxonomic proposals to the International Committee on Taxonomy of Viruses (2008)". Archives of Virology. 154 (7): 1181–88. doi:10.1007/s00705-009-0400-2. PMC 7086627. PMID 19495937.
- ^ Racaniello VR, Baltimore D (novembar 1981). "Cloned poliovirus complementary DNA is infectious in mammalian cells". Science. 214 (4523): 916–19. Bibcode:1981Sci...214..916R. doi:10.1126/science.6272391. PMID 6272391.
- ^ Cello J, Paul AV, Wimmer E (august 2002). "Chemical synthesis of poliovirus cDNA: generation of infectious virus in the absence of natural template". Science. 297 (5583): 1016–18. Bibcode:2002Sci...297.1016C. doi:10.1126/science.1072266. PMID 12114528. S2CID 5810309.
- ^ Couzin J (juli 2002). "Virology. Active poliovirus baked from scratch". Science. 297 (5579): 174–75. doi:10.1126/science.297.5579.174b. PMID 12114601. S2CID 83531627.
- ^ Brown MC, Dobrikova EY, Dobrikov MI, Walton RW, Gemberling SL, Nair SK, et al. (novembar 2014). "Oncolytic polio virotherapy of cancer". Cancer. 120 (21): 3277–86. doi:10.1002/cncr.28862. PMC 4205207. PMID 24939611.
Vanjski linkovi
[uredi | uredi izvor]- ICTVdb virus classification 2006
- Home of Picornaviruses (latest updates of species, serotypes, & proposed changes) Arhivirano 4. 11. 2010. na Wayback Machine
- Goodsell D. "Poliovirus and Rhinovirus". August 2001 Molecule of the Month. Arhivirano s originala, 3. 3. 2011. Pristupljeno 11. 4. 2022.
- 3D macromolecular structures of the Poliovirus archived in the EM Data Bank(EMDB)
- "Human poliovirus 1". NCBI Taxonomy Browser. 12080.
- "Human poliovirus 3". NCBI Taxonomy Browser. 12086.
